ユビキチン化は、細胞内で不要となったタンパク質を選別・分解する重要な制御機構です。本記事では、ユビキチン化の流れや関与するE1〜E3酵素、そしてデグロン(N末デグロン、PEST配列、D-box)による分解シグナルについて、例を交えてわかりやすく解説します。
Contents
タンパク質分解の目印、ユビキチン
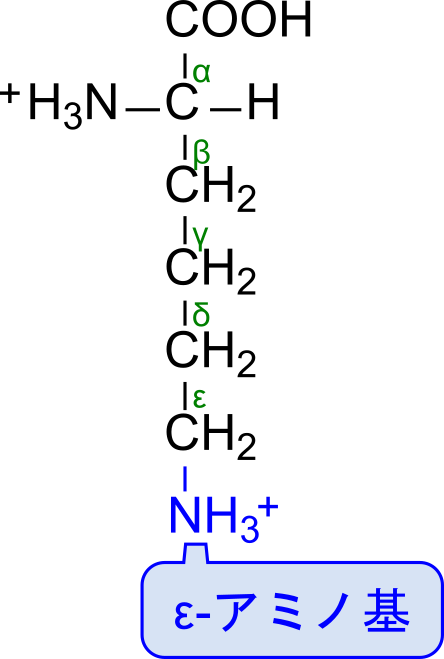
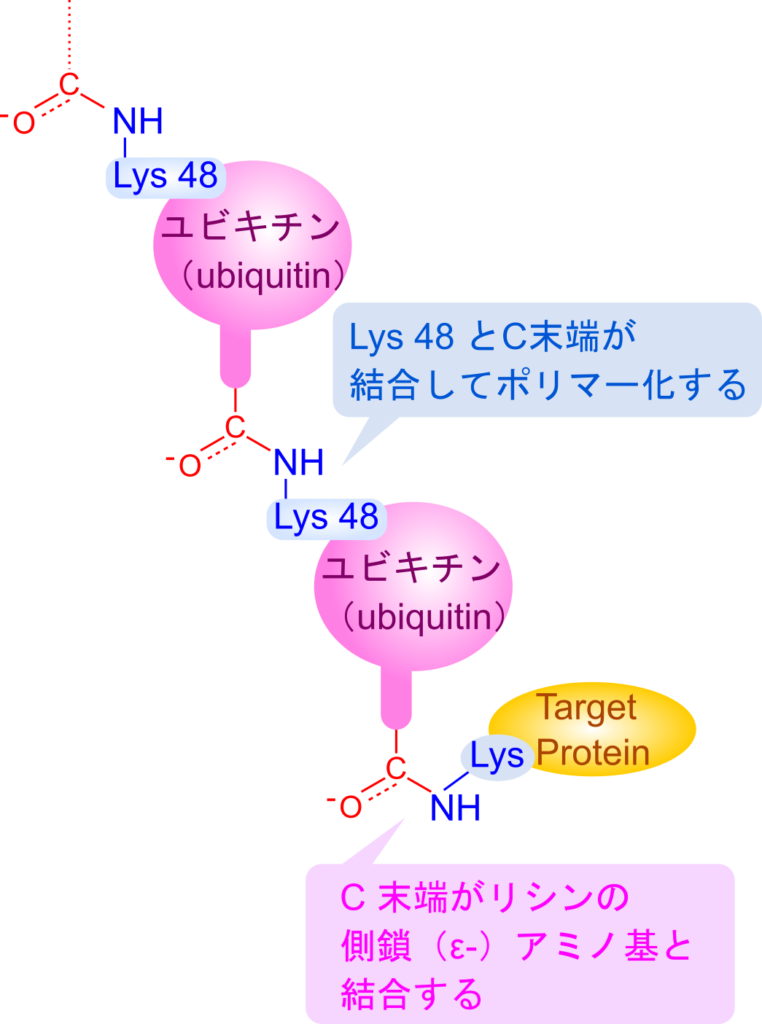
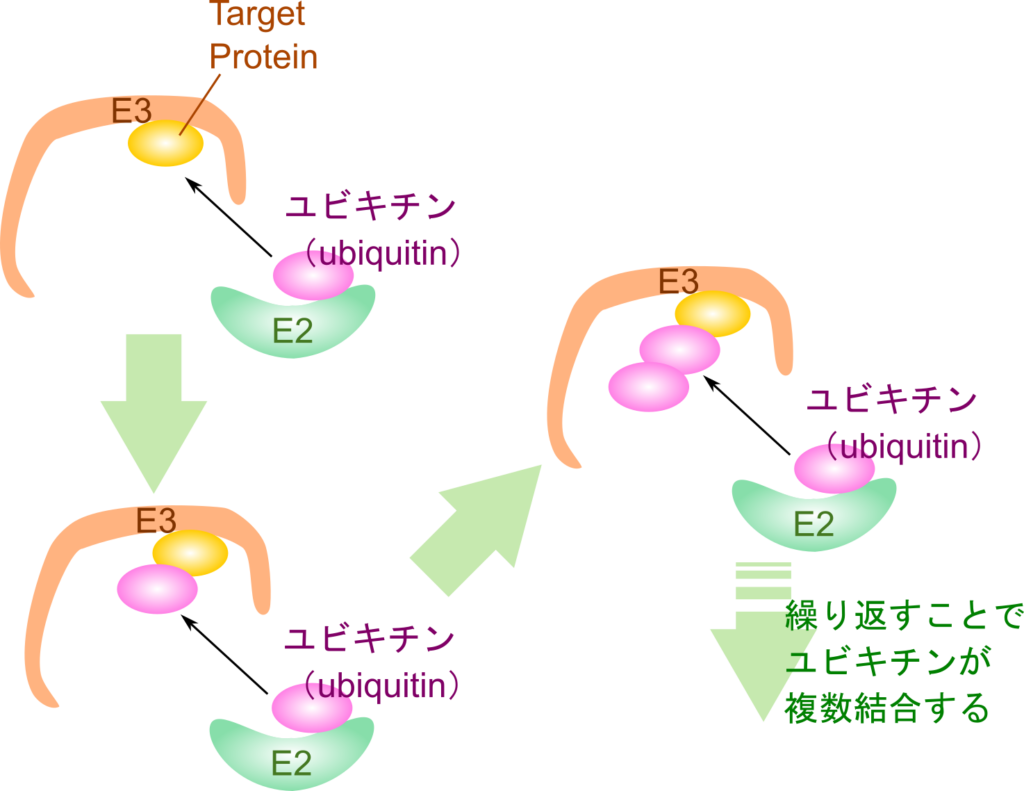
寿命を迎えて分解するべきタイミングになるとタンパク質にユビキチンが結合します(この過程はユビキチン化(Ubiquitination)と呼ばれます)。ヒトのユビキチンは 76 アミノ酸残基から成るタンパク質で、C 末端側(カルボキシル基で終端している末端)が標的タンパク質のリシン残基の側鎖(ε-)アミノ基とペプチド結合を形成します(この結合はリシンの側鎖に結合するので主鎖から見ると枝分かれしているような関係になります。このことからイソペプチド結合(isopeptide bond)と呼ばれます)。のちに解説しますが、ユビキチンは通常、標的タンパク質に対して複数個が結合することで、より有効なシグナルとして機能します。標的タンパク質に結合したユビキチンに新たなユビキチンを結合させるときは、前のユビキチンの 48 番目のリシン残基と新たなユビキチンの C 末端がペプチド結合を形成してつながります。この過程を繰り返し、複数のユビキチンがポリマーを形成します(図1)。
標的タンパク質のユビキチン化メカニズム
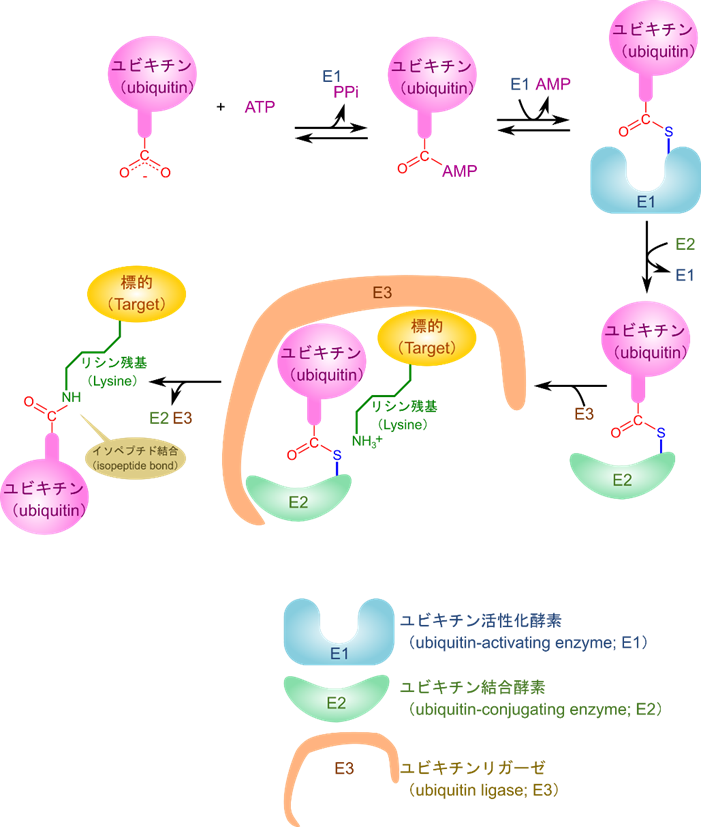
この反応はユビキチン活性化酵素(E1)、ユビキチン結合酵素(E2)、ユビキチン-タンパク質リガーゼ(E3)の連携と ATP の消費によって達成されます。
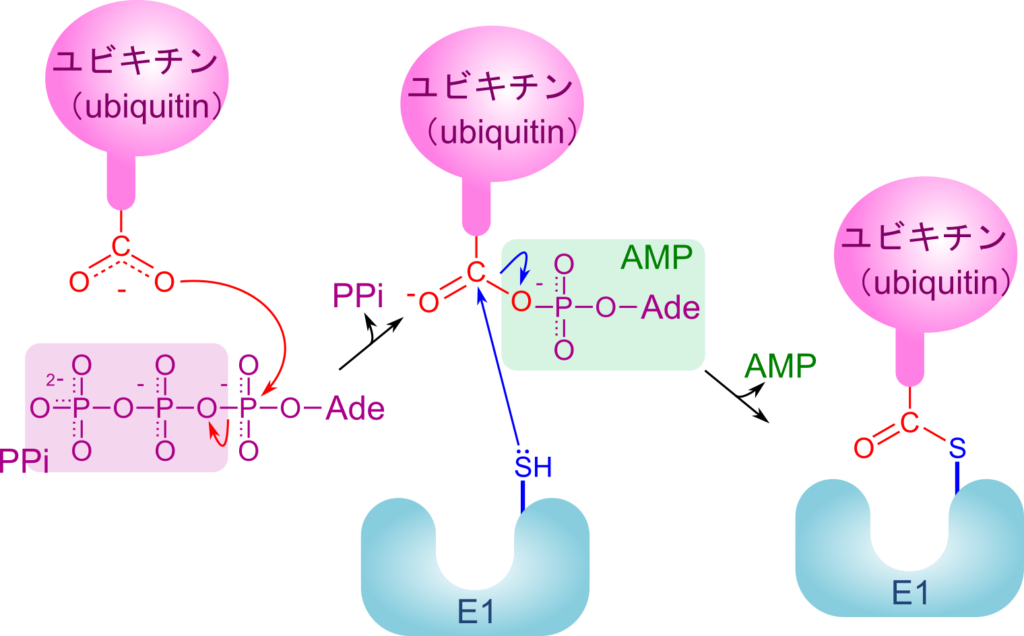
この反応ではまず、ユビキチンの C 末端に AMP が結合します。この時、ATP からピロリン酸(PPi)が脱離して、アデノシンがリン酸基を介してカルボキシル基に結合します。この反応は E1 が触媒することで進行します(アシルアデニル化;acyl-adenylation)。このアシルアデニル化によりユビキチンの C 末端が活性化され、続く反応が起こりやすくなります。次に、E1 上の(システイン上の)チオール基がカルボニル基の炭素を攻撃して結合し、AMP が脱離します。こうしてユビキチンが E1 に結合しました(図2)。
その後チオエステル(-CO-S-)のチオール側が E1 から E2 に交換されます。このチオールの交換は E2 自身が触媒して進行します。最終的に、E3 が標的タンパク質のリシン残基の ε アミノ基とユビキチンの C 末端のペプチド結合形成を触媒し、E2 が遊離してユビキチン化が終了します。
この反応は E3 が標的タンパク質と結合したまま連続して繰り返されることがあります。この場合、新たなユビキチンは前のユビキチンの 48 番目のリシン残基の側鎖アミノ基に転位されていきます(上記の反応で標的タンパク質をそのままユビキチン化標的タンパク質に入れ替えたイメージです)。その結果、複数個のユビキチンが標的タンパク質に結合させられます。このような複数のユビキチン(ポリユビキチン)はより効果的なシグナルとして機能します。このようにユビキチンは分解するべきタンパク質の目印として機能しますが、一方でユビキチン化するべきタンパク質は厳密に制御されていなければなりません。そうでなければ、機能を維持するべきタンパク質が分解されてしまう原因となってしまいます。では、ユビキチン化を受けるか否かはどのように制御されているのでしょうか?次の項目でユビキチン化の制御についてみていきましょう。
ユビキチン化はタンパク質の配列により制御されている

ユビキチンが結合するべきか否かを決定するのはデグロン(degron)と呼ばれるアミノ酸配列です。デグロンにはN末デグロン(N-degron)、サイクリン破壊ボックス(cyclin destruction box)、PEST 配列(プロリン(P)、グルタミン酸(E)、セリン(S)、トレオニン(T)を含む配列でそれぞれの一字表記にちなんで PEST 配列と呼ばれます)など、いくつかの種類が知られています。
N末デグロン(N-degron)

タンパク質の半減期はアミノ基末端(-NH2 が露出する側の末端;N 末端とも言います)がどんなアミノ酸残基なのかに大きく依存します。この性質を N 末端則(N end rule)といい、このようなシグナルを N 末デグロン(N-degron)といいます。
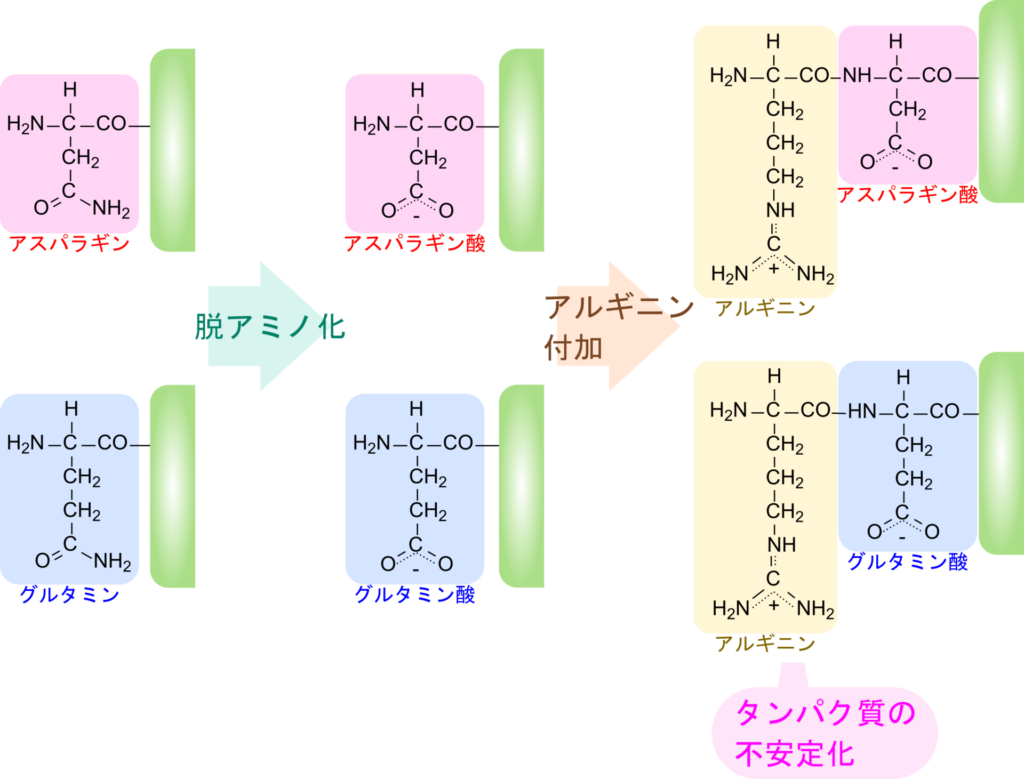
N 末端にアラニンやメチオニンを持つタンパク質は半減期は 20 時間以上となり、比較的安定です。これはユビキチン化されにくいために寿命が延びていることを意味しています。一方で、アルギニンやロイシンのようなアミノ酸残基の場合には半減期はかなり短期化され、2~30分とタンパク質は不安定化して分解されやすくなります。このような場合にはタンパク質はユビキチン化されやすく、そのためタンパク質は速やかに分解されるようになります(図5)。N末端がアスパラギン酸やグルタミン酸の場合にはアルギニンが付与されることで修飾され、タンパク質は不安定化されます。一方で、アスパラギンやグルタミンの場合には脱アミノ化されてアスパラギン酸やグルタミン酸に分解され、アルギニン付加による修飾を受けることで、アルギニンを有する N 末デグロンとなりますが、上述の通り、アルギニンはタンパク質を不安定化させる N 末デグロンです。したがって、タンパク質は速やかに分解されるようになります。このように、アスパラギン、アスパラギン酸、グルタミン、グルタミン酸の四つのアミノ酸残基では修飾されることによりタンパク質を不安定化するシグナルとなります(図6)。
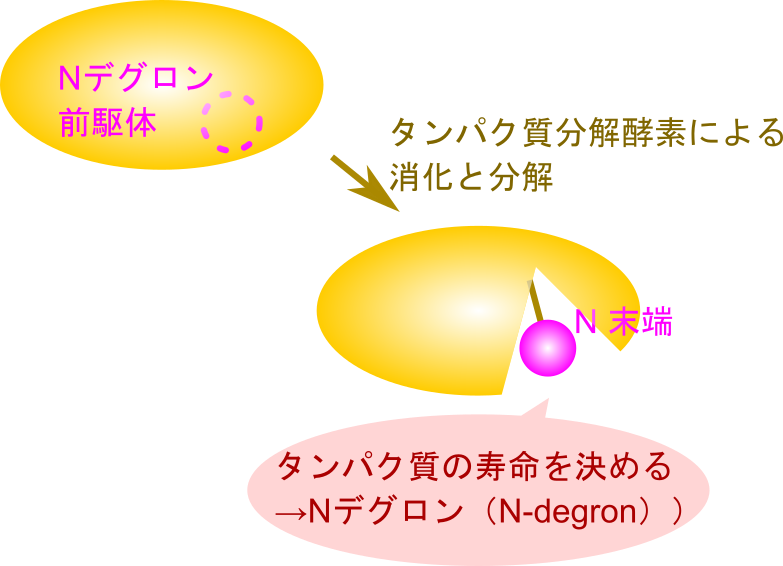
ところで、タンパク質を不安定化させるような配列(デグロン)は、通常タンパク質内部に隠れています。このため、本来機能すべきタンパク質が誤って分解されることはありません。一方で、タンパク質の中には、特定のタンパク質分解酵素によって切断されることで活性化されたり、逆に分解されてその機能を終えるものもあります。こうした過程は、タンパク質がその役割を適切に果たす上で重要です。このように、タンパク質が分解酵素によって切断されていく過程で、内部に埋もれていたN末端のデグロンが露出し、ユビキチン化が誘導されることがあります。このとき、切断前の状態でタンパク質内部に存在していたデグロン配列は、「N末デグロン前駆体(N-degron precursor)」と呼ばれます(念のため補足しますが、デグロンとはタンパク質中に存在する特定のアミノ酸配列のことであり、N末デグロン前駆体もタンパク質の特定の配列を指します。したがって、タンパク質全体ではなく、部分配列を指す点に注意してください)。このようにして、N末デグロンは適切なタイミングで露出し、タンパク質の分解が制御される仕組みとなっています(図7)。
破壊ボックス(destruction box;D-box)
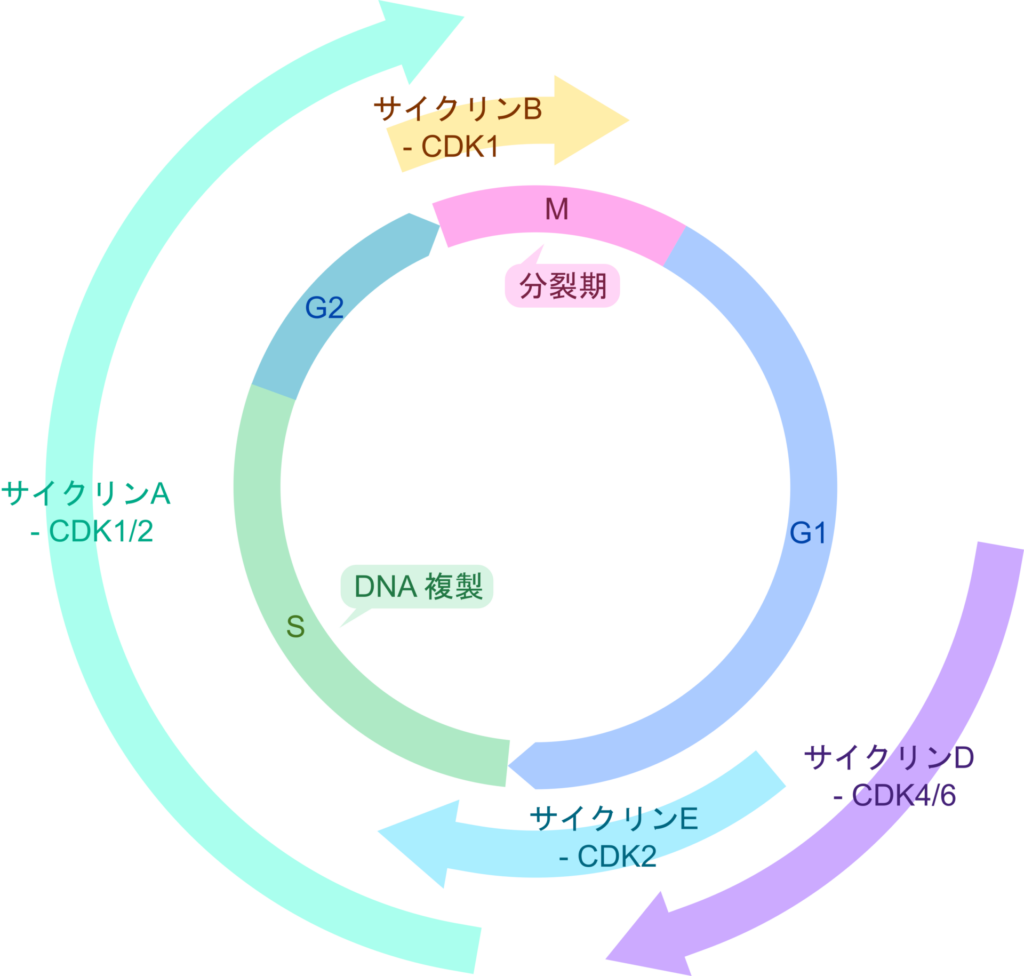
細胞周期の各段階には細胞周期を調整するサイクリン-CDK 複合体が用意されており、これらのタンパク質が適切なタイミングで機能を発揮することで細胞は複製と分裂を繰り返していきます。
Destruction box(D-box)は、RXXLという特徴的なアミノ酸配列(このようにある種の機能に関与する配列のことをモチーフ(Motif)といいます)を含む構造で、ユビキチン依存的分解を誘導する「デグロン」の一種です。D-boxを持つ代表的なタンパク質には、細胞周期のM期に関与するサイクリンBやセキュリンがあります。
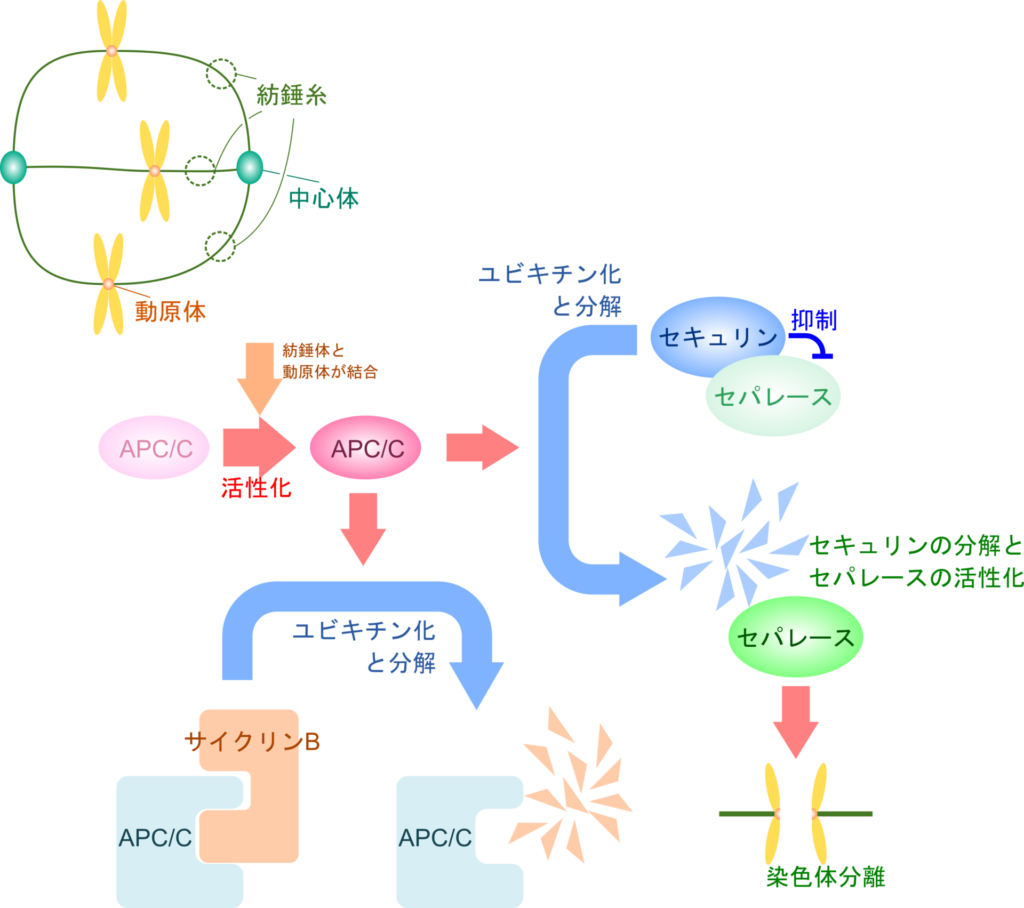
細胞周期の M 期では、細胞分裂が起こります。サイクリン B-CDK1 複合体は細胞を M 期へ進行させる役割を担っていますが(図8)、M 期後期になると役割を終えたサイクリン B は内包する destruction box (D-box) を ユビキチンリガーゼ(E3)である APC/C(anaphase promoting complex/cyclosome)が認識することでユビキチン化が起こります。APC/C は紡錘体が染色体の動原体と正しく結合するまでは活性を抑制されているため、M 期後期に至るまで APC/C が機能することはありません。一方で、APC/C はセキュリンにも作用します。セキュリンはセパレースと複合体を形成している因子で、セパレースの機能を抑制しています。セパレースは染色体の分離を誘導するタンパク質ですので、セキュリンの機能により適切な時期まで染色体分離が起こることを抑制していると考えられています。動原体と紡錘体が結合すると APC/C は活性化され、セキュリンの D-box を認識してユビキチン化が誘導されて分解されることでセキュリンが分解され、セパレースの活性が誘導された結果、染色体の分離が起こります。こうして、動原体と紡錘体の結合から染色体分離が順序良く起こるようになっています。このように、D-box や APC/C によるタンパク質のユビキチン化と分解が細胞周期を適切に進行させることに関与しています(図9)。
PEST 配列(PEST sequence)
PEST 配列はプロリン(P)、グルタミン酸(E)、セリン(S)、トレオニン(T)〜に富むアミノ酸配列で構成される領域で、タンパク質の分解を促すデグロン(分解シグナル)として機能します。PEST 配列を持つタンパク質は迅速に分解されることが知られており、リン酸化を受けることがタンパク質をユビキチン化とその後のプロテアソーム分解のトリガーとなることが知られています。代表的な例は NFκB(炎症応答、免疫応答、アポトーシス、腫瘍形成に関与する遺伝子の発現を調節する転写因子)の阻害因子である IκBα(PEST 配列:MLPESEDEESY)、サイクリンD1 (PEST 配列:PEPEPESESEETTT;C 末端付近)などがあります。
練習問題
ユビキチンが標的タンパク質に結合するアミノ酸はどれ?
E1酵素の役割として正しいものはどれ?
E2酵素の役割として正しいものはどれ?
次のうち、E3ユビキチンリガーゼの役割として正しいものはどれ?
次のうち、デグロンとして機能する配列はどれ?
今回の記事では、タンパク質の寿命をコントロールする「ユビキチン化」と、それを導くシグナルである「デグロン」について解説しました。ユビキチン化は、単なる不要タンパク質の処理にとどまらず、細胞内の重要な反応を開始させる“スイッチ”としての役割も果たしています。特に、N末デグロン、D-box、PEST配列といった多様なデグロンの機能を知ることで、「いつ」「どこで」「どのように」タンパク質が分解されるのかという、生命活動の精緻な制御メカニズムに触れることができます。
この記事は、ユビキチン化の基本原理にとどまらず、それが細胞の秩序や調和にどのように寄与しているかという“生物学的意義”にも踏み込んでいます。ぜひ、必要に応じて何度でも読み返し、理解を深めるきっかけにしていただければ幸いです。
参考文献
- Jeremy M. Berg, John L. Tymoczko, Gregory J. Gatto Jr., Lubert Stryer著、入村達郎、岡山博人、清水孝雄、中野徹訳 (2018). ストライヤー生化学 第8版. 東京化学同人. pp.633-662
- D. Voet, J. G. Voet, C. W. Pratt 著、田宮信雄、八木達彦、遠藤斗志也、吉久徹訳 (2017) ボート 基礎生化学 第5版. 東京化学同人. pp.477-513
- Park, J. S., Lee, J.-Y., Nguyen, Y. T. K., Kang, N.-W., Oh, E. K., Jang, D. M., … Han, B. W. (2020). Structural Analyses on the Deamidation of N-Terminal Asn in the Human N-Degron Pathway. Biomolecules, 10(1), 163. https://doi.org/10.3390/biom10010163
- Varshavsky, A. (2024). N-degron pathways. Proceedings of the National Academy of Sciences, 121(39). https://doi.org/10.1073/pnas.2408697121
- Wang, H., Piatkov, K. I., Brower, C. S., & Varshavsky, A. (2009). Glutamine-Specific N-Terminal Amidase, a Component of the N-End Rule Pathway. Molecular Cell, 34(6), 686–695. https://doi.org/10.1016/j.molcel.2009.04.032
- Alfieri, C., Zhang, S., & Barford, D. (2017). Visualizing the complex functions and mechanisms of the anaphase promoting complex/cyclosome (APC/C). Open Biology, 7(11). https://doi.org/10.1098/rsob.170204
- Lu, D., Hsiao, J. Y., Davey, N. E., Van Voorhis, V. A., Foster, S. A., Tang, C., & Morgan, D. O. (2014). Multiple mechanisms determine the order of APC/C substrate degradation in mitosis. Journal of Cell Biology, 207(1), 23–39. https://doi.org/10.1083/jcb.201402041
- Sue, S. C., & Dyson, H. J. (2009). Interaction of the IκBα C-terminal PEST Sequence with NF-κB: Insights into the Inhibition of NF-κB DNA Binding by IκBα. Journal of Molecular Biology, 388(4), 824–838. https://doi.org/10.1016/j.jmb.2009.03.048
- Wang, X., Peng, H., Huang, Y., Kong, W., Cui, Q., Du, J., & Jin, H. (2020, November 5). Post-translational Modifications of IκBα: The State of the Art. Frontiers in Cell and Developmental Biology. Frontiers Media S.A. https://doi.org/10.3389/fcell.2020.574706
- Montalto, F. I., & De Amicis, F. (2020). Cyclin D1 in Cancer: A Molecular Connection for Cell Cycle Control, Adhesion and Invasion in Tumor and Stroma. Cells, 9(12), 2648. https://doi.org/10.3390/cells9122648
- Wang, J., Su, W., Zhang, T., Zhang, S., Lei, H., Ma, F., … Di, C. (2023, April 1). Aberrant Cyclin D1 splicing in cancer: from molecular mechanism to therapeutic modulation. Cell Death and Disease. Springer Nature. https://doi.org/10.1038/s41419-023-05763-7